This is a deep learning model that was developed to predict PPAR-gamma ligands (agonists and antagonists). Here are some specs:
- Accuracy (5-fold cross-validation): 99.74%
- True Positive Rate: 99% --> ligand를 positive라고 본다.
- False Positive Rate: 0.5%
- Probably that a PPAR-gamma ligand will be predicted to be a ligand: P(ligand | predict: ligand): 89%
- Probability that a non-ligand will be predicted to be a ligand: P(\not{ligand} | predict: ligand): 11%.

[[756, 6],
[ 8, 754]]
true positive rate = (756/(756+6)) : 실제 ligand(0)중 ligand(0)로 예측된 비율
false positive rate = (8/(8+754)) : 실제 (not_ligand)중에 (ligand)로 예측된 비율
https://github.com/DataSciBurgoon/ppar-gamma-model/blob/master/ppar-gamma_ligand_mlp.ipynb
DataSciBurgoon/ppar-gamma-model
PPAR-gamma deep learning model. Contribute to DataSciBurgoon/ppar-gamma-model development by creating an account on GitHub.
github.com
질문1 P(ligand | predict: ligand)과 precision의 차이가 무엇일까?
https://darkpgmr.tistory.com/62
베이즈 정리, ML과 MAP, 그리고 영상처리
고등학교 수학에서 조건부 확률이라는 걸 배운다. 그런데 그게 나중에 가면 베이지안 확률이라는 이름으로 불리면서 사람을 엄청 햇갈리게 한다. 1. 베이즈 정리 베이즈 정리(Bayes's theorem) 또는
darkpgmr.tistory.com
drive.google.com/file/d/1kV5iy2EnNqZVo2ozS_kVMCSy4OQM-gAJ/view?usp=sharing
ppar-gamma_ligand_mlp.ipynb
Colaboratory notebook
drive.google.com
Now I'm going to do some chemical structure comparisons graphically to see if I can identify some structures that ligands might share. This isn't an exhaustive search, and one isn't possible. However, this may help to illuminate some structures that are shared by known ligands. In this case, I'm going to be focusing on the known ligands pirinixic acid, liothyronine, oxaprozin, lansoprazole, romazarit, and linoleic acid.
ligand들이 공유할 수 있는 몇가지 구조를 식별하기 위해, 몇몇의 화학물질 구조를 비교를 graphically하게 수행하도록 하겠습니다.
완전한 search는 불가능하다. 그러나 일부 공유하는 구조를 밝히는데 도움이 될 수 있다.
pirinixic acid, :작용제
liothyronine, :길항제
oxaprozin,
lansoprazole,
romazarit,
linoleic acid.
Pirinixic acid CC1=C(C(=CC=C1)NC2=CC(=NC(=N2)SCC(=O)O)Cl)C ligand
liothyronine C1=CC(=C(C=C1OC2=C(C=C(C=C2I)CC(C(=O)O)N)I)I)O ligand
oxaprozin C1=CC=C(C=C1)C2=C(OC(=N2)CCC(=O)O)C3=CC=CC=C3 ligand
lansoprazole CC1=C(C=CN=C1CS(=O)C2=NC3=CC=CC=C3N2)OCC(F)(F)F ligand
Romazarit CC1=C(OC(=N1)C2=CC=C(C=C2)Cl)COC(C)(C)C(=O)O ligand
linoleic acid CCCCCC=CCC=CCCCCCCCC(=O)O ligand

Of all the ligands, linoleic acid is the one that is most at risk of looking like a non-ligand; however, it still has a very strong probability at 0.057 (recall that scores closer to 0 are more likely ligands, while scores closer to 1 are more likely non-ligands. Let's examine how the linoleic acid structure compares to many of the others.
Just among this small random sampling of chemicals, a few characteristics are beginning to emerge. Keep in mind, these compounds represent agonists and antagonists. The reference compound, pirinixic acid, oxaprozin, and romazarit are agonists. Liothyronine and lansoprazole are antagonists.
What one sees is that aromatic groups, carboxylic acids, and imines tend to be shared amongst the group of ligands. In addition, oxazole rings, ethers, and amines tend to not be shared amongst the PPARg ligands, based on comparison to the prototypic pirinixic acid structure.
모든 ligand중 리놀레산(linoleic)은 non-ligand처럼 보일 확률이 높지만 여전치 0.057이라는 강력한 수치를 가진다.(0에 가까울수록 liand일 가능성이 높고 1에 가까울수록 non-ligand일 가능성이 높다)
리놀레산 구조가 다른 물질들의 구조와 어떻게 비교되는지 살펴보도록하겠다.
무작위로 화학물질을 샘플링해본 결과 몇가지 특성이 나타났다.
샘플링한 화합물들은 작용제와 길항제이다.
pirinixic acid, oxaprozin, and romazarit are agonists(작용제).
Liothyronine and lansoprazole are antagonists(길항제.
우리가 본 것은 방향족 그룹, 카르복실 산 , imines이 ligand그룹간에 공유되는 경향이 있다는 것이다.
ligand : 단백질이나 화학물질과 결합하는 아이들.
oxazole rings, ethers, and amines는 PPARg 리간드에서 공유되지 않는 경향이 있다.
prototypic pirinixic acid 구조와 비교하여
PPARg 리간드 그룹간에 공유되는 경향이 있는애들
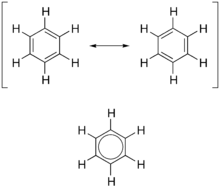
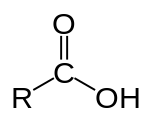
PPARg 리간드에서 공유되지 않는 경향이 있는애들(prototypic pirinixic acid 에 비해)



FingerFrint란 무엇인가?
mols = []
fps = []
#get molecules and then get fingerprints from those
for index, row in dataframe.iterrows():
mol = Chem.MolFromSmiles(row['SMILES'])
fp = AllChem.GetMorganFingerprintAsBitVect(mol, 2)
mols.append(mol)
fps.append(fp)
#Convert the RDKit vectors into numpy arrays
#Based on: http://www.rdkit.org/docs/Cookbook.html#using-scikit-learn-with-rdkit
np_fps = []
for fp in fps:
arr = numpy.zeros((1,))
DataStructs.ConvertToNumpyArray(fp, arr)
np_fps.append(arr)
np_fps_array = numpy.array(np_fps)